使用AI工具MONAI和RAPIDS,快速分析全玻片图像
2023年07月14日 由 Alex 发表
861611
0
数字病理切片扫描仪生成大量图像。玻片通常以40倍的放大倍率扫描,产生十亿像素的图像。压缩可以将每张幻灯片的文件大小减少到1或2GB,但是移动、保存、加载和查看这些数据量仍然具有挑战性。要以全分辨率观看典型的整张幻灯片图像,需要一台网球场大小的显示器。
像组织病理学一样,基因组学和显微镜学都可以很容易地产生TB级的数据。一些用例涉及多个模式。将这些数据转换为更易于管理的大小通常涉及逐步转换,直到只保留最突出的特性。本文探讨了一些数据细化可能完成的方法,使用的分析类型,以及MONAI和RAPIDS等工具如何解锁有意义的见解。它以典型的数字组织病理学图像为例,因为这些图像现在在全球范围内的常规临床环境中使用。
MONAI是一套开源、免费提供的协作框架,旨在加速医学成像领域的研究和临床协作。RAPIDS是一套开源软件库,用于在GPU上构建端到端数据科学和分析管道。RAPIDS cuCIM是一种用于多维图像的计算机视觉处理软件库,可以加速MONAI的成像,cuDF库可以帮助完成工作流程所需的数据转换。
前面已经展示了cuCIM如何加速整个幻灯片图像的加载。
但是管道的其余部分(可能包括图像预处理、推理、后处理、可视化和分析)怎么办呢?越来越多的仪器捕获各种数据,包括多光谱图像,以及遗传和蛋白质组学数据,所有这些都提出了类似的挑战。
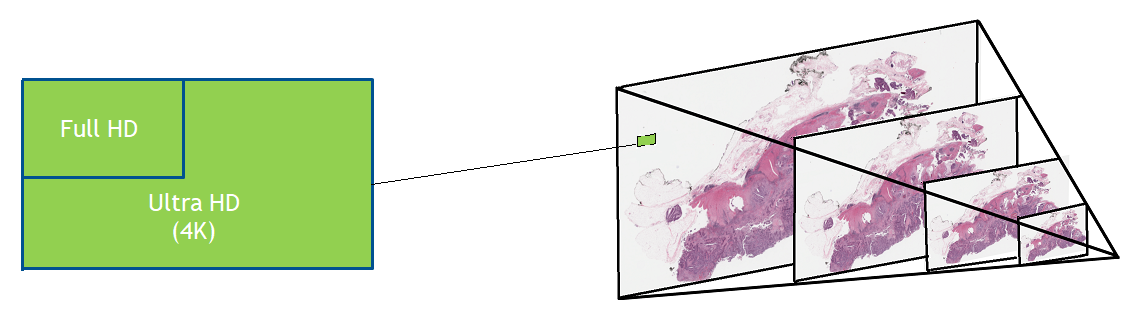
癌症等疾病起源于细胞核,其大小只有5-20微米。为了辨别不同的细胞亚型,形状、颜色、内部纹理和模式需要对病理学家可见。这需要非常大的图像。
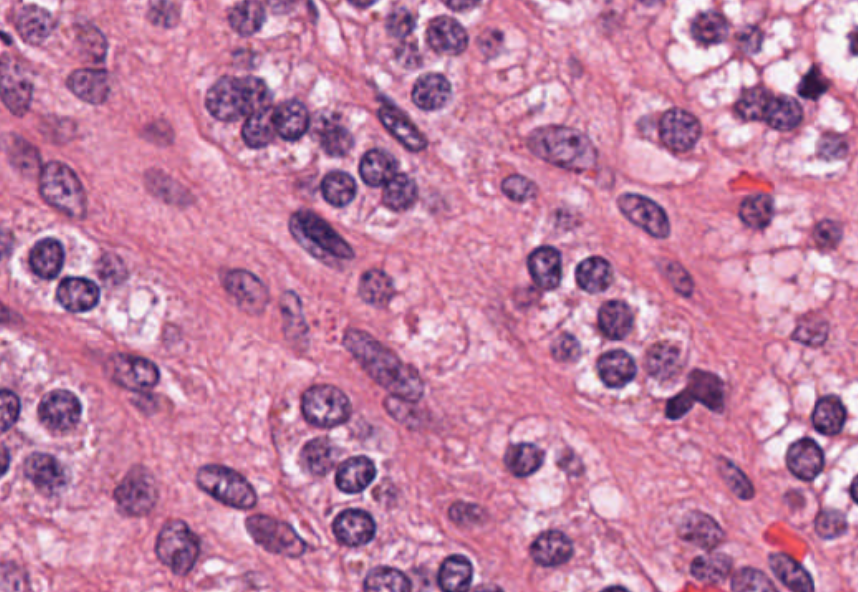
考虑到2D深度学习算法(如DenseNet)的普通输入大小通常在200 x 200像素左右,高分辨率图像需要拆分为一个切片(可能为10万个)。
载玻片制备和组织染色过程可能需要数小时。虽然低延迟推断结果的价值是最小的,但分析仍然必须跟上数字扫描仪的获取率,以防止积压。因此,吞吐量至关重要。提高吞吐量的方法是更快地处理图像或同时计算多个图像。
数据科学家和开发人员已经考虑了许多方法来使问题更容易处理。考虑到图像的大小和病理学家必须进行诊断的有限时间,没有实用的方法可以以全分辨率查看每个像素。
相反,他们以较低的分辨率查看图像,然后放大他们认为可能包含感兴趣特征的区域。他们通常可以在查看全分辨率图像的1-2%后做出诊断。在某些方面,这就像一个侦探在犯罪现场:大部分现场都是不相关的,结论通常取决于一两根纤维或指纹,这些纤维或指纹提供了关键信息。
与人类不同,人工智能和机器学习(ML)无法丢弃图像中98-99%的像素,因为担心它们可能会错过一些关键细节。这在未来是可能的,但需要相当大的信任和证据来证明它是安全的。
在这方面,目前的算法平等地对待所有输入像素。随后,各种算法机制可能会给它们分配或多或少的权重(Attention, Max-Pooling, Bias和Weights),但最初它们都具有相同的影响预测的潜力。
这不仅给组织病理学处理管道带来了巨大的计算负担,而且还需要在磁盘、CPU和GPU之间移动大量数据。大多数组织病理学玻片包含空白、冗余信息和噪声。可以利用这些属性来减少提取重要信息所需的实际计算。
例如,病理学家可以通过统计相关区域内的某些细胞类型来对疾病进行分类。要做到这一点,该算法必须将像素强度值转换为带有相关细胞类型标签的核质心数组。这样,计算区域内的细胞计数就非常简单了。可以通过多种方式将整个幻灯片图像过滤到特定任务的基本元素。一些例子可能包括:
对于这两种方法中的任何一种,MONAI都提供了许多模型和培训管道,您可以根据自己的需要进行定制。大多数都是通用的,足以适应您的数据的特定需求(例如通道和维度的数量),但有几个是特定于数字病理学的。
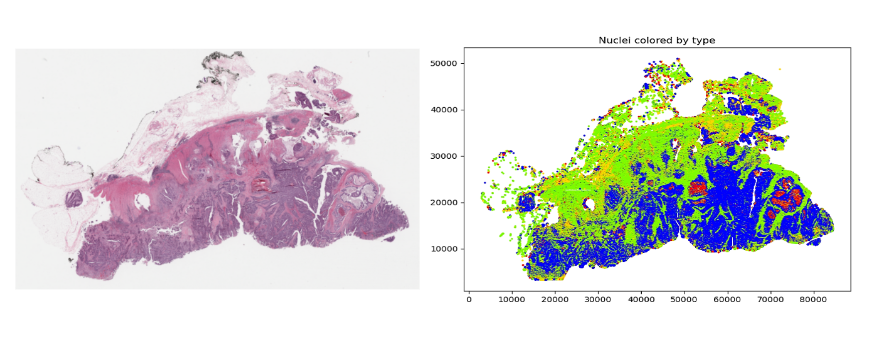
导出这些特征后,它们就可以用于分析。然而,即使在这种类型的降维之后,仍然可能有许多特征需要分析。例如,图3显示了具有数十万个核的图像(最初为100K x 60K RGB像素)。即使为每个64 x 64贴图生成嵌入,也可能导致一张幻灯片产生数百万个数据点。
这就是RAPIDS可以提供帮助的地方。使用Python的GPU加速数据科学的开源库套件包括涵盖一系列常见活动的工具,例如ML,图形分析,ETL和可视化。有一些底层技术,比如CuPy,可以让不同的操作访问GPU内存中的相同数据,而不必复制或重构底层数据。这是RAPIDS如此快速的主要原因之一。
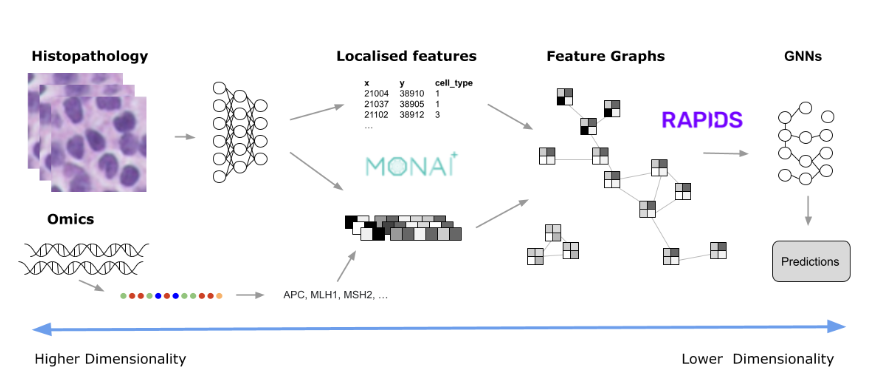
开发人员的主要交互工具之一是CUDA加速数据帧(cuDF)。数据以表格格式呈现,可以使用cuDF API与类似熊猫一样的命令进行过滤和操作,使其易于采用。然后将这些数据帧用作许多其他RAPIDS工具的输入。
例如,假设要从所有原子核创建一个图形,将每个原子核与特定半径内最近的相邻原子核连接起来。为此,需要向cuGraph API提供一个数据帧,其中的列表示每个图边的源节点和目标节点(具有可选的权重)。要生成此列表,可以使用cuML最近邻搜索功能。同样,只需提供一个列出所有核坐标的数据帧,cuML就会完成所有繁重的工作。
请注意,在默认情况下,计算的距离是欧氏距离,为了节省不必要的计算,它们是平方值。其次,该算法默认使用启发式算法。如果需要实际值,可以指定可选参数。无论哪种方式,在GPU上计算都是非常快的。
接下来,将距离和索引数据帧合并为一个数据帧。要做到这一点,首先需要为距离列分配唯一的名称:
每行必须对应于图中的一条边,因此数据帧需要为每个最近的邻居拆分为一行。然后可以将列重命名为“源”、“目标”和“距离”。
要消除一定距离以外的所有相邻要素,请使用以下过滤器:
此时,您可以省去“距离”列,除非图形中的边需要加权。然后创建图形本身:
有了图形之后,就可以进行标准的图形分析操作了。三角形计数是长度为3的循环数。图的k核是包含k度以上节点的最大子图:
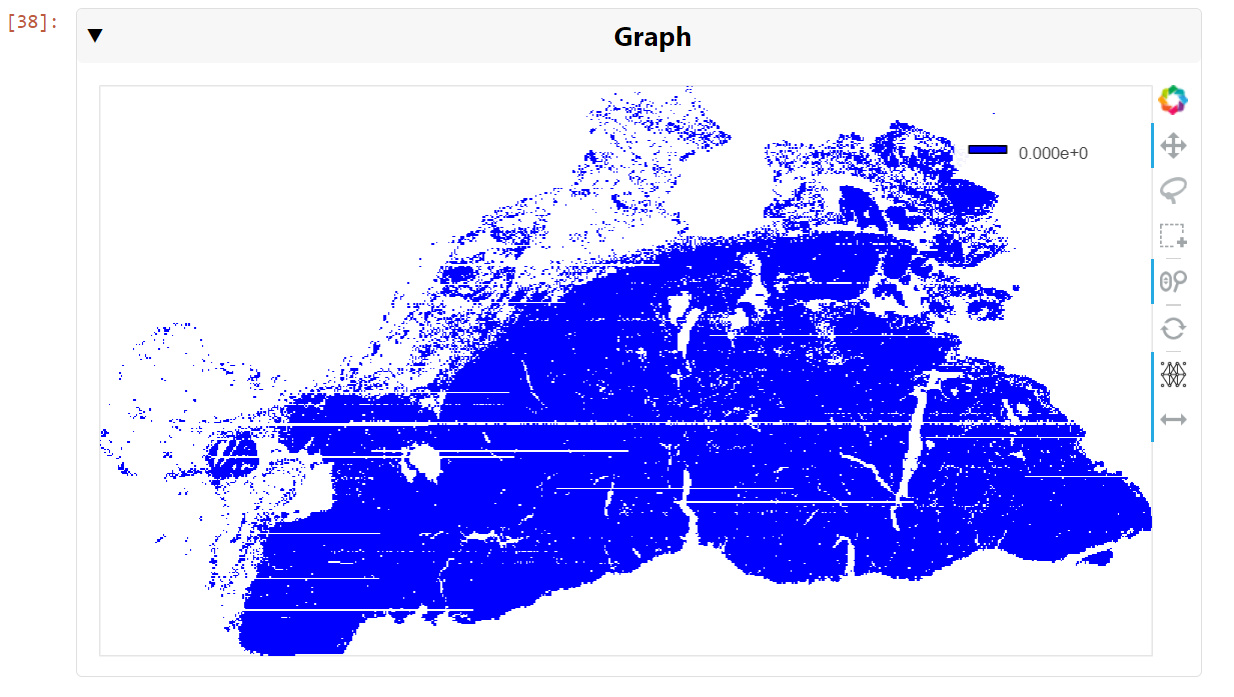
也可以将图形可视化,即使它可能包含数十万条边。使用现代GPU,可以实时查看和导航图形。要生成这样的可视化效果,请使用cuXFilter:
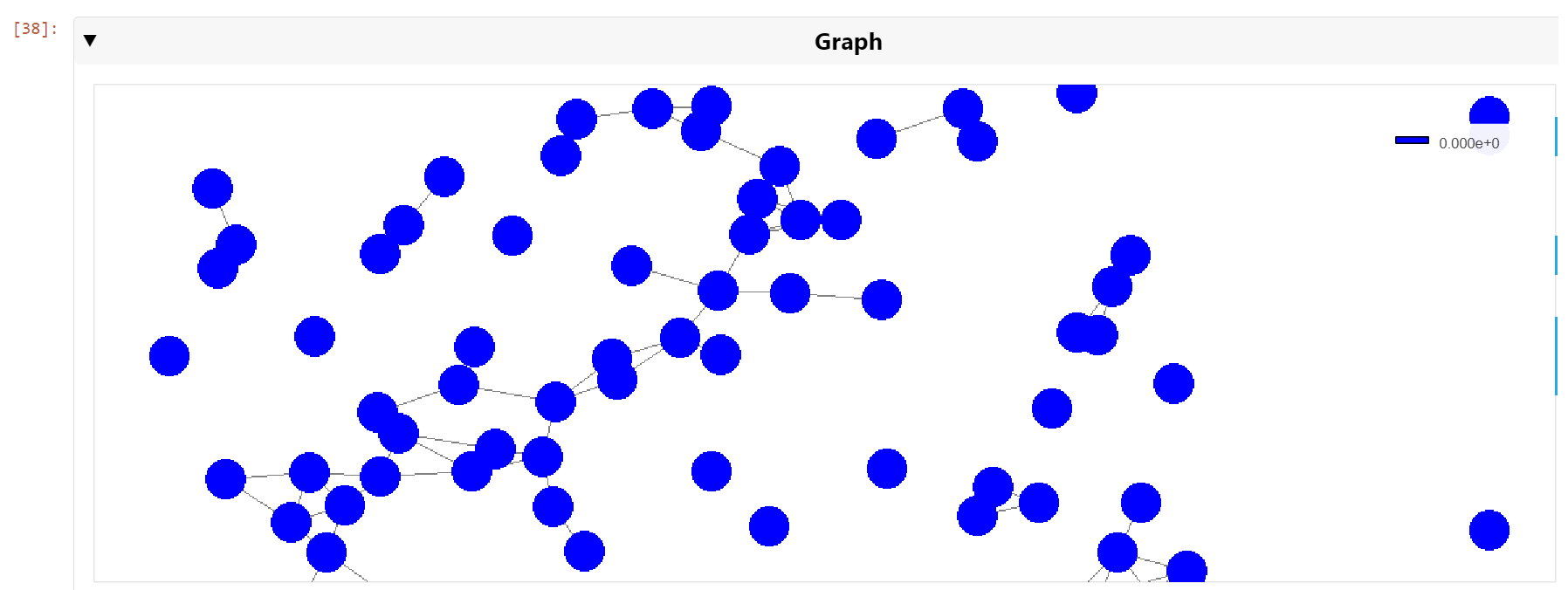
然后,您可以平移并缩小到细胞核级别,以查看最近邻居的集群(图6)。
从原始像素中提取见解可能是困难和耗时的。一些强大的工具和技术可以应用于大图像问题,以提供近乎实时的分析,甚至是最具挑战性的数据。除了ML功能外,GPU加速工具(如RAPIDS)还提供强大的可视化功能,有助于破译基于DL的方法产生的计算特征。本文描述了一组端到端的工具,它可以使用DL、ML Graph和GNN方法来摄取、预处理、推断、后处理、绘图。
来源:https://developer.nvidia.com/blog/whole-slide-image-analysis-in-real-time-with-monai-and-rapids/
像组织病理学一样,基因组学和显微镜学都可以很容易地产生TB级的数据。一些用例涉及多个模式。将这些数据转换为更易于管理的大小通常涉及逐步转换,直到只保留最突出的特性。本文探讨了一些数据细化可能完成的方法,使用的分析类型,以及MONAI和RAPIDS等工具如何解锁有意义的见解。它以典型的数字组织病理学图像为例,因为这些图像现在在全球范围内的常规临床环境中使用。
MONAI是一套开源、免费提供的协作框架,旨在加速医学成像领域的研究和临床协作。RAPIDS是一套开源软件库,用于在GPU上构建端到端数据科学和分析管道。RAPIDS cuCIM是一种用于多维图像的计算机视觉处理软件库,可以加速MONAI的成像,cuDF库可以帮助完成工作流程所需的数据转换。
管理整个幻灯片图像数据
前面已经展示了cuCIM如何加速整个幻灯片图像的加载。
但是管道的其余部分(可能包括图像预处理、推理、后处理、可视化和分析)怎么办呢?越来越多的仪器捕获各种数据,包括多光谱图像,以及遗传和蛋白质组学数据,所有这些都提出了类似的挑战。

图1
癌症等疾病起源于细胞核,其大小只有5-20微米。为了辨别不同的细胞亚型,形状、颜色、内部纹理和模式需要对病理学家可见。这需要非常大的图像。

图2
考虑到2D深度学习算法(如DenseNet)的普通输入大小通常在200 x 200像素左右,高分辨率图像需要拆分为一个切片(可能为10万个)。
载玻片制备和组织染色过程可能需要数小时。虽然低延迟推断结果的价值是最小的,但分析仍然必须跟上数字扫描仪的获取率,以防止积压。因此,吞吐量至关重要。提高吞吐量的方法是更快地处理图像或同时计算多个图像。
潜在解决方案
数据科学家和开发人员已经考虑了许多方法来使问题更容易处理。考虑到图像的大小和病理学家必须进行诊断的有限时间,没有实用的方法可以以全分辨率查看每个像素。
相反,他们以较低的分辨率查看图像,然后放大他们认为可能包含感兴趣特征的区域。他们通常可以在查看全分辨率图像的1-2%后做出诊断。在某些方面,这就像一个侦探在犯罪现场:大部分现场都是不相关的,结论通常取决于一两根纤维或指纹,这些纤维或指纹提供了关键信息。

图3
与人类不同,人工智能和机器学习(ML)无法丢弃图像中98-99%的像素,因为担心它们可能会错过一些关键细节。这在未来是可能的,但需要相当大的信任和证据来证明它是安全的。
在这方面,目前的算法平等地对待所有输入像素。随后,各种算法机制可能会给它们分配或多或少的权重(Attention, Max-Pooling, Bias和Weights),但最初它们都具有相同的影响预测的潜力。
这不仅给组织病理学处理管道带来了巨大的计算负担,而且还需要在磁盘、CPU和GPU之间移动大量数据。大多数组织病理学玻片包含空白、冗余信息和噪声。可以利用这些属性来减少提取重要信息所需的实际计算。
例如,病理学家可以通过统计相关区域内的某些细胞类型来对疾病进行分类。要做到这一点,该算法必须将像素强度值转换为带有相关细胞类型标签的核质心数组。这样,计算区域内的细胞计数就非常简单了。可以通过多种方式将整个幻灯片图像过滤到特定任务的基本元素。一些例子可能包括:
1. 使用无监督方法学习一组图像特征,例如训练变分自编码器,将图像块编码为小嵌入。
2. 定位所有感兴趣的特征(例如,细胞核),并且仅使用此信息专用模型(如HoVerNet)来获取指标。
MONAI和RAPIDS
对于这两种方法中的任何一种,MONAI都提供了许多模型和培训管道,您可以根据自己的需要进行定制。大多数都是通用的,足以适应您的数据的特定需求(例如通道和维度的数量),但有几个是特定于数字病理学的。
导出这些特征后,它们就可以用于分析。然而,即使在这种类型的降维之后,仍然可能有许多特征需要分析。例如,图3显示了具有数十万个核的图像(最初为100K x 60K RGB像素)。即使为每个64 x 64贴图生成嵌入,也可能导致一张幻灯片产生数百万个数据点。
这就是RAPIDS可以提供帮助的地方。使用Python的GPU加速数据科学的开源库套件包括涵盖一系列常见活动的工具,例如ML,图形分析,ETL和可视化。有一些底层技术,比如CuPy,可以让不同的操作访问GPU内存中的相同数据,而不必复制或重构底层数据。这是RAPIDS如此快速的主要原因之一。

图4
开发人员的主要交互工具之一是CUDA加速数据帧(cuDF)。数据以表格格式呈现,可以使用cuDF API与类似熊猫一样的命令进行过滤和操作,使其易于采用。然后将这些数据帧用作许多其他RAPIDS工具的输入。
例如,假设要从所有原子核创建一个图形,将每个原子核与特定半径内最近的相邻原子核连接起来。为此,需要向cuGraph API提供一个数据帧,其中的列表示每个图边的源节点和目标节点(具有可选的权重)。要生成此列表,可以使用cuML最近邻搜索功能。同样,只需提供一个列出所有核坐标的数据帧,cuML就会完成所有繁重的工作。
from cuml.neighbors import NearestNeighbors
knn = NearestNeighbors()
knn.fit(cdf)
distances, indices = knn.kneighbors(cdf, 5)
请注意,在默认情况下,计算的距离是欧氏距离,为了节省不必要的计算,它们是平方值。其次,该算法默认使用启发式算法。如果需要实际值,可以指定可选参数。无论哪种方式,在GPU上计算都是非常快的。
接下来,将距离和索引数据帧合并为一个数据帧。要做到这一点,首先需要为距离列分配唯一的名称:
distances.columns=['ix2','d1','d2','d3','d4']
all_cols = cudf.concat(
[indices[[1,2,3,4]], distances[['d1','d2','d3','d4']]],
axis=1)
每行必须对应于图中的一条边,因此数据帧需要为每个最近的邻居拆分为一行。然后可以将列重命名为“源”、“目标”和“距离”。
all_cols['index1'] = all_cols.index
c1 = all_cols[['index1',1,'d1']]
c1.columns=['source','target','distance']
c2 = all_cols[['index1',2,'d2']]
c2.columns=['source','target','distance']
c3 = all_cols[['index1',3,'d3']]
c3.columns=['source','target','distance']
c4 = all_cols[['index1',4,'d4']]
c4.columns=['source','target','distance']
edges = cudf.concat([c1,c2,c3,c4])
edges = edges.reset_index()
edges = edges[['source','target','distance']]
要消除一定距离以外的所有相邻要素,请使用以下过滤器:
distance_threshold = 15
edges = edges.loc[edges["distance"] < distance_threshold**2]
此时,您可以省去“距离”列,除非图形中的边需要加权。然后创建图形本身:
cell_graph = cugraph.Graph()
cell_graph.from_cudf_edgelist(edges,source='source', destination='target', edge_attr='distance', renumber=True)
有了图形之后,就可以进行标准的图形分析操作了。三角形计数是长度为3的循环数。图的k核是包含k度以上节点的最大子图:
count = cugraph.triangle_count(cell_graph)
coreno = cugraph.core_number(cell_graph)
也可以将图形可视化,即使它可能包含数十万条边。使用现代GPU,可以实时查看和导航图形。要生成这样的可视化效果,请使用cuXFilter:
nodes = tiles_xy_cdf
nodes['vertex']=nodes.index
nodes.columns=['x','y','vertex']
cux_df = fdf.load_graph((nodes, edge_df))
chart0 = cfc.graph(
edge_color_palette=['gray', 'black'],
timeout=200,
node_aggregate_fn='mean',
node_pixel_shade_type='linear',
edge_render_type='direct',#other option available -> 'curved', edge_transparency=0.5)
d = cux_df.dashboard([chart0], layout=clo.double_feature)
chart0.view()

图5
然后,您可以平移并缩小到细胞核级别,以查看最近邻居的集群(图6)。

图6
结论
从原始像素中提取见解可能是困难和耗时的。一些强大的工具和技术可以应用于大图像问题,以提供近乎实时的分析,甚至是最具挑战性的数据。除了ML功能外,GPU加速工具(如RAPIDS)还提供强大的可视化功能,有助于破译基于DL的方法产生的计算特征。本文描述了一组端到端的工具,它可以使用DL、ML Graph和GNN方法来摄取、预处理、推断、后处理、绘图。
来源:https://developer.nvidia.com/blog/whole-slide-image-analysis-in-real-time-with-monai-and-rapids/

欢迎关注ATYUN官方公众号
商务合作及内容投稿请联系邮箱:bd@atyun.com
热门企业
热门职位
写评论取消
回复取消